Перейти к:
Молекулярно-генетические особенности антибиотикорезистентности Pseudomonas aeruginosa
https://doi.org/10.34215/1609-1175-2024-2-47-50
Аннотация
Цель: провести генетический анализ штаммов Pseudomonas aeruginosa для выяснения изменения их антибиотикорезистентности с 2012 по 2023 год.
Материалы и методы. 174 штамма P. aeruginosa, выделенные у пациентов многопрофильного стационара г. Владивостока, разделенные на три группы в зависимости от года выделения: в 2012, 2019 (до пандемии коронавирусной инфекции) и 2023 гг. (после пандемии). Для проведения полимеразной цепной реакции использовали гены антибиотикорезистентности OXA23, ISAbaI, IMP, VIM, TEM, NDM.
Результаты. Частота выявления всех изучаемых генетических детерминант антибиотикорезистентности возрастала с течением времени. Наиболее высокий рост выявлен за период пандемии (с 2019 по 2023 г.) для ISAbaI (с 69 до 100%), VIM (с 50 до 70%), IMP (с 28 до 48%) и TEM (с 24 до 96%).
Заключение. Выявлено появление и распространение среди P. aeruginosa важных генетических детерминант антибиотикорезистентности, в том числе NDM-1. Это отражает общие тенденции в популяции и говорит о все более широком распространении детерминант антибиотикорезистентности, и соответственно, о повышении общего уровня устойчивости к антимикробным препаратам у возбудителей ИСМП.
Ключевые слова
Для цитирования:
Скурихина Ю.Е., Зайцева Е.А., Сараговец А.А. Молекулярно-генетические особенности антибиотикорезистентности Pseudomonas aeruginosa. Тихоокеанский медицинский журнал. 2024;(2):47-50. https://doi.org/10.34215/1609-1175-2024-2-47-50
For citation:
Skurikhina Yu.E., Zaitseva E.A., Saragovets A.A. Molecular genetic features of Pseudomonas aeruginosa antibiotic resistance. Pacific Medical Journal. 2024;(2):47-50. (In Russ.) https://doi.org/10.34215/1609-1175-2024-2-47-50
Инфекции, связанные с оказанием медицинской помощи (ИСМП), являются актуальной проблемой в современном здравоохранении. Их эндемический уровень представляет серьезную угрозу для пациентов и медицинского персонала. Одна из наиболее распространенных этиологических причин ИСМП – Pseudomonas aeruginosa. Этот микроорганизм вызывает широкий спектр заболеваний, включая инфекции мочевыводящих путей, хирургические инфекции, инфекции системы кровообращения и госпитальные пневмонии. В мире и в России в современный период отмечается постоянный рост частоты инфекций, вызванных P. aeruginosa [1][2].
P. aeruginosa имеет множество первичных и приобретенных факторов антибиотикорезистентности, среди них – металло-β-лактамазы (МБЛ), оксациллиназы, другие ферменты, которые разрушают антибиотики [3]. Инфекции, вызванные P. aeruginosa, трудно поддаются лечению и требуют применения новых методов и подходов для борьбы с ними. Медицинское учреждение, в котором возникает вспышка ИСМП, сталкивается с серьезными проблемами лечения пациента, существенным увеличением расходов и возможным отрицательным влиянием на имидж и репутацию.
Пандемия COVID-19 привела к повышению распространенности ИСМП, вызванных условно-патогенными возбудителями, в том числе P. aeruginosa. Это обусловлено увеличением числа пациентов с ослабленным иммунитетом, изменением структуры заболеваемости и ограничением доступа к стандартным методам диагностики и лечения. Также изменилась и антибиотикорезистентность многих микроорганизмов в связи с повсеместным ростом применения антибиотиков широкого спектра действия [4][5].
Цель исследования состояла в генетическом анализе штаммов P. aeruginosa для выяснения их молекулярного разнообразия и изменения антибиотикорезистентности с 2012 по 2023 год.
Материалы и методы
Материалом послужили 174 штамма P. aeruginosa, выделенные у пациентов многопрофильного стационара г. Владивостока в 2012 (55 штаммов), 2019 (55 штаммов) и 2023 году (64 штамма).
В качестве маркеров генотипов был выбран ряд генов, кодирующих важные факторы антибиотикорезистентности. Для молекулярно-генетического анализа использовали гены OXA23, ISAbaI, IMP, VIM, TEM, NDM. Последовательности специфических праймеров приведены в таблице.
Таблица
ДНК-праймеры, использованные для ПЦР-анализа штаммов P. aeruginosa
Праймер | Нуклеотидные последовательности | Размер фрагмента, п. н. | Автор |
OXA-23 | F: 5’-ACTTGCTATGTGGTTGCTTCTC-3’ R: 5’-TGTCAAGCTCTTAAATATTCAGC-3’ | 791 | [6] |
ISAba1 | F: 5’-CACGAATGCAGAAGTTG-3’ R: 5’-CGACGAATACTATGACAC-3’ | 549 | [6] |
TEM | F: 5’-ATAAAATTCTTGAAGACGAAA-3’ R: 5’-GACAGTTACCAATGCTTAATCA-3’ | 1080 | [7] |
NDM | F: 5’-GGTTTGGCGATCTGGTTTTC-3’ R: 5’-CGGAATGGCTCATCACGATC-3’ | 621 | [8] |
IMP | F: 5’-GTTTATGTTCATACWTCG-3’ R: 5’-GGTTTAAYAAAACAACCAC-3’ | 432 | [9] |
VIM | F: 5’-TTTGGTCGCATATCGCAACG-3’ R: 5’-CCATTCAGCCAGATCGGCAT-3’ | 500 | [9] |
Молекулярно-генетический анализ проводили с использованием полимеразной цепной реакции (ПЦР) на термоциклере С 1000TM (ThermalCycler, Bio-Rad, USA). ДНК выделяли с помощью набора для выделения геномной ДНК из бактериальных клеток (diaGene, «Диаэм», Россия).
Рабочая смесь содержала: 1 ед. HS Taq ДНК-полимеразы («Евроген», Россия), 1×буфер (10×: 600 mМ Tris-HCl, 250 mМ KCl, 15mМ MgCl2 , 100 mM 2-меркаптоэтанол, 1% Тритон-X-100, pH 8,5), 0,8 мМ смеси дезоксинуклеозидтрифосфатов (0,2 мМ каждого), 0,25 μМ каждого праймера и 50 нг ДНК. ПЦР проводили в условиях, рекомендованных авторами (табл.). Продукты амплификации анализировали с помощью электрофореза в 1–1,5% агарозном геле (Agarose Biotechnology Grade, «Amresco», USA) с окраской ДНК этидиум бромидом. Размер амплифицированных фрагментов определяли по стандартным маркерам молекулярной массы фрагментов ДНК, кратных 100 либо 50 парам нуклеотидов (п.н.) («СибЭнзим», «Евроген», Россия).
Результаты исследования
Первоначально все исследуемые штаммы разделили на три группы в зависимости от года выделения: в 2012 г., до коронавирусной инфекции (2019) и в период после этой инфекции (2023 г.). Частота встречаемости генов МБЛ IMP, VIM, TEM была следующей: в выборке 1 ген blaIMP определялся в 19%, в выборке 2 – в 28%, в выборке 3 – в 48% случаев.
Ген blaVIM – в 45, 50 и 70, blaTEM – в 4, 24 и 96% соответственно. У штаммов из выборки 1 ген МБЛ NDM-1 не встречался, в выборке 2 – уже в 32%, в выборке 3 – в 33% случаев.
Частота встречаемости гена оксациллиназы OXA-23 в выборках 1, 2, 3 была 32, 36, 37% соответственно.
Ген ISAba1 встречался в 60, 69 и 100% случаев в исследуемых группах штаммов (рис.).
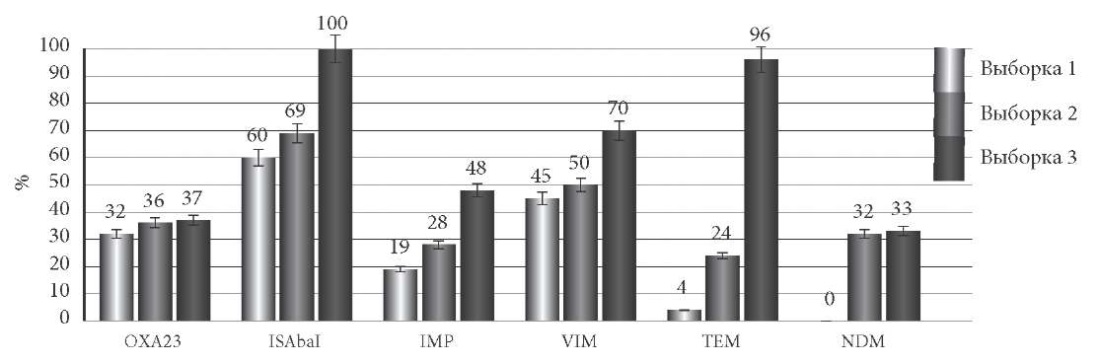
Рис. Частота встречаемости генов резистентности у штаммов P. aeruginosa:
в выборке 1 (2012 г.), выборке 2 (2019 г.), выборке 3 (2023 г.).
Наиболее высокий рост частоты встречаемости генов выявлен для ISAbaI, IMP, VIM и TEM, превышающий таковой между анализируемыми выборками в несколько раз по сравнению с предыдущим семилетним временным разрывом (2012 и 2019 гг.).
Обсуждение полученных данных
В настоящее время резистентность микроорганизмов к антимикробным препаратам рассматривается как изменение генома бактерий в результате мутации и последующей селекции наиболее удачных вариантов.
Существует по крайней мере девять разных видов приобретенных МБЛ. Среди них IMP, VIM, TEM и NDM считаются наиболее важными с точки зрения распространенности и клинической значимости. Выявление генетических детерминант β-лактамаз – один из самых актуальных методов определения комплексной устойчивости к антибиотикам. Согласно мировым исследованиям, их распространенность составляет 10–95% [7-9]. МБЛ TEM – наиболее часто встречающаяся бета-лактамаза у грамотрицательных бактерий, обеспечивает устойчивость в том числе к сульбактаму, потенциальная терапевтическая эффективность которого выше, чем у других ингибиторов β-лактамаз (клавулановой кислоты и тазобактама) [7]. В соответствии с полученными данными наблюдается распространение этих МБЛ в популяциях P. aeruginosa с течением времени, особенно TEM (на 20% за период 2012–2019 гг., и еще на 72% – по сравнению с доковидным 2019 г.). Частота встречаемости IMP и VIM также возросла на 29 и 25% соответственно в целом и на 20% к послековидному 2023 г.
NDM-1 – еще одна МБЛ, эпидемиологически значимый маркер резистентных штаммов. Бактерии, несущие этот ген, резистентны к β-лактамным антибиотикам, включая пиперациллин/тазобактам, азтреонам, все карбапенемы, цефтазидим и цефепим, и сохраняют чувствительность только к тигециклину и колистину [8]. В популяции штаммов, выделенных в 2012 году, данный ген не встречался. В 2019 году его обнаружили уже у 32% штаммов. Интересно, что в постковидном периоде частота его встречаемости практически не изменилась.
Среди генетических детерминант антибиотикорезистентности описано несколько классов оксациллиназ, наибольшую роль среди них играет OXA-23, чаще всего у мультирезистентных штаммов и связана с развитием резистентности к карбапенемам [6]. Инсерционный элемент ISAba1 содержит промоторы, которые участвуют в экспрессии генов, кодирующих OXA-23. ISAba1 можно считать микробиологически значимым маркером резистентных госпитальных штаммов [6v7]. В последний исследуемый период (2019–2023 гг.) он встречался у 100% исследуемых штаммов, тогда как в 2019 году – у 69%. Но выявляемость OXA-23 повысилась лишь на 1%, при этом резких изменений за весь исследуемый период (2012–2023 гг.) не наблюдалось.
Следует отметить увеличение распространенности всех исследуемых генов. Особенно в постковидный период по сравнению с предыдущими возросла частота ISAba1 (с 60 до 100%), VIM (с 45 до 70%) и TEM (с 4 до 96%). Это отражает общие тенденции в популяциях микроорганизмов – возбудителей ИСМП, в том числе P. aeruginosa [1][10][11], и говорит о все более широком распространении детерминант антибиотикорезистентности и, соответственно, о повышении общего уровня устойчивости к антимикробным препаратам у возбудителей ИСМП.
Заключение
Бактерии P. aeruginosa содержат генетические детерминанты антибиотикорезистентности, которые обрели широкое распространение в период пандемии COVID-19. Рутинные микробиологические методы не позволяют полноценно оценить спектр возбудителей у одного пациента, степень родства госпитальных штаммов, уровень антибиотикорезистентности и другие важные показатели. Для эффективной борьбы с ИСМП необходимо широкое использование современных генетических методов для мониторинга возбудителей ИСМП и антибиотикорезистентности, это позволит отразить реальную эпидемиологическую ситуацию и обеспечить эффективность профилактических мер.
Конфликт интересов: авторы декларируют отсутствие явных и потенциальных конфликтов интересов, связанных с публикацией настоящей статьи.
Источник финансирования: авторы заявляют о финансировании проведенного исследования из собственных средств.
Участие авторов:
Концепция и дизайн исследования – СЮЕ, ЗЕА
Сбор и обработка материала – СЮЕ, САА
Написание текста – СЮЕ, ЗЕА
Редактирование – ЗЕА
Список литературы
1. Serretiello E, Manente R, Dell’Annunziata F, Folliero V, Iervolino D, Casolaro V, Perrella A, Santoro E, Galdiero M, Capunzo M, Franci G, Boccia G. Antimicrobial resistance in Pseudomonas aeruginosa before and during the COVID-19 pandemic. Microorganisms. 2023;11(8):1918. doi: 10.3390/microorganisms11081918
2. Воропаева Н.М., Немченко У.М., Григорова Е.В., Белькова Н.Л., Чемезова Н.Н., Савилов Е.Д. Этиологическая структура инфекций, связанных с оказанием медицинской помощи, и антибиотикорезистетность основных возбудителей инфекций. Эпидемиология и вакцинопрофилактика. 2023;22(1);68–73. doi: 10.31631/2073-3046-2023-22-1-68-73
3. Hong D, Bae IK, Jang I. Epidemiology and Characteristics of Metallo-β-Lactamase-Producing Pseudomonas aeruginosa. Infect Chemother. 2015; 47(2):81–97. doi: 10.3947/ic.2015.47.2.81
4. Гомон Ю.М., Колбин А.С., Буданов Д.С. Динамика потребления антимикробных препаратов резерва в стационарах России: влияние пандемии COVID-19. Антибиотики и Химиотерапия. 2023;68(5–6):62–8. doi: 10.37489/0235-2990-2023-68-5-6-62-68
5. Потапов А.Ф., Шамаева С.Х., Иванова А.А., Семенова С.В. Микрофлора ран и резистентность к антибиотикам у пострадавших с термической травмой. Тихоокеанский медицинский журнал, 2023;1(91);81–5 doi: 10.34215/1609-1175-2023-1-81-85
6. Carvalho KR, D’Alincourt Carvalho-Assef AP, dos Santos LG. Occurrence of blaOXA-23 gene in imipenem-susceptible Acinetobacter baumannii. Mem Inst Oswaldo Cruz, Rio de Janeiro. 2011;106(4):505–6. doi: 10.1590/S0074-02762011000400020
7. Mohajeri P, Rostami Z, Farahani A, Norozi B. Distribution of ESBL producing uropathogenic Escherichia coli and carriage of selected β-lactamase genes in hospital and community isolates in west of Iran. Ann Tropic Med Pub Health. 2014;7(5):219–22. doi: 10.4103/1755-6783.154823
8. Fomda BA, Khan A, Zahoor D. NDM-1 (New Delhi metallo beta lactamase-1) producing Gram-negative bacilli: emergence & clinical implications. Indian J Med Res. 2014;140(5):672–8.
9. Amudhan MS, Sekar U, Kamalanathan A, Balaraman S. bla(IMP) and bla(VIM) mediated carbapenem resistance in Pseudomonas and Acinetobacter species in India. J Infect Dev Ctries. 2012;6(11):757–62. doi: 10.3855/jidc.2268
10. Перфильева Д.Ю., Мирошниченко А.Г., Куликов Е.С., Бойк ов В.А., Нестерович С.В., Перфильев В.Ю. Внутрибольничные инфекции: взгляд на проблему в условиях глобальной угрозы антибиотикорезистентности (обзор). Сибирский журнал клинической и экспериментальной медицины. 2024;39(1):28–37. doi: 10.29001/2073-8552-2024-39-1-28-37
11. Samarkos M, Skouloudi M, Anastasopoulou A, Markogiannakis A. Restricted antimicrobial prescribing in an area of highly prevalent antimicrobial resistance. Infect. Dis. Now. 2021;51(6):526–31. doi: 10.1016/j.idnow.2021.05.002
Об авторах
Ю. Е. СкурихинаРоссия
Скурихина Юлия Евгеньевна – канд. мед. наук, заведующий кафедрой эпидемиологии и военной эпидемиологии
690002, Владивосток, пр-т Острякова, 2
Е. А. Зайцева
Россия
Владивосток
А. А. Сараговец
Россия
Владивосток
Рецензия
Для цитирования:
Скурихина Ю.Е., Зайцева Е.А., Сараговец А.А. Молекулярно-генетические особенности антибиотикорезистентности Pseudomonas aeruginosa. Тихоокеанский медицинский журнал. 2024;(2):47-50. https://doi.org/10.34215/1609-1175-2024-2-47-50
For citation:
Skurikhina Yu.E., Zaitseva E.A., Saragovets A.A. Molecular genetic features of Pseudomonas aeruginosa antibiotic resistance. Pacific Medical Journal. 2024;(2):47-50. (In Russ.) https://doi.org/10.34215/1609-1175-2024-2-47-50





































